Какое число мутаций может кардинально изменить укладку белка?
advertisement
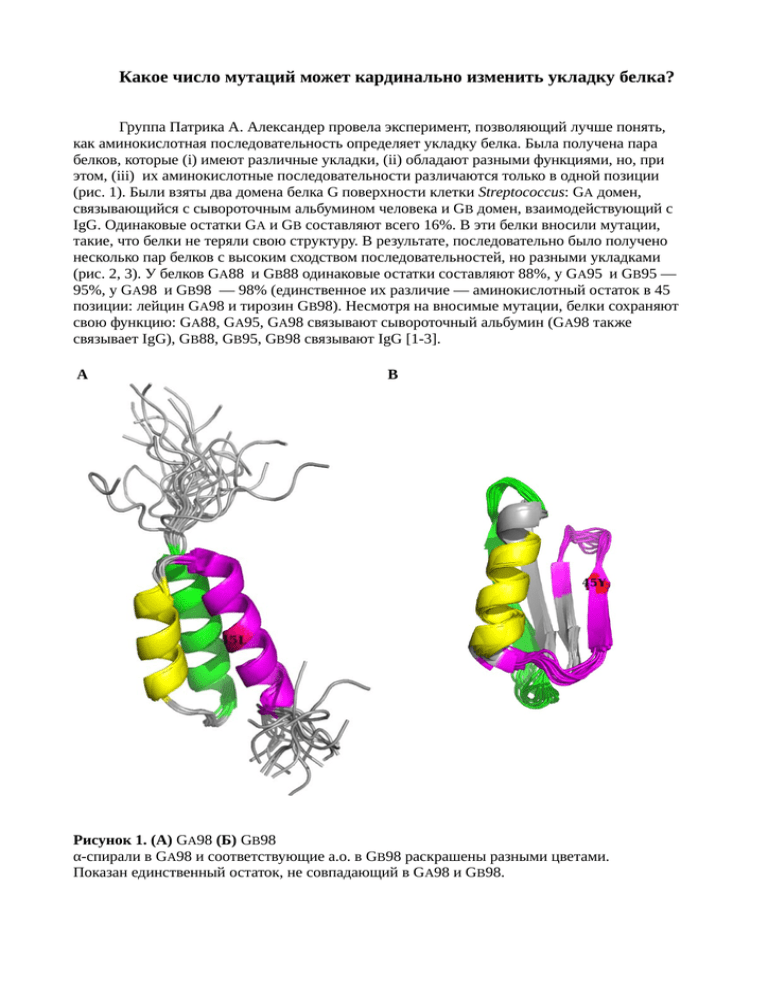
Какое число мутаций может кардинально изменить укладку белка? Группа Патрика А. Александер провела эксперимент, позволяющий лучше понять, как аминокислотная последовательность определяет укладку белка. Была получена пара белков, которые (i) имеют различные укладки, (ii) обладают разными функциями, но, при этом, (iii) их аминокислотные последовательности различаются только в одной позиции (рис. 1). Были взяты два домена белка G поверхности клетки Streptococcus: GA домен, связывающийся с сывороточным альбумином человека и GB домен, взаимодействующий с IgG. Одинаковые остатки GA и GB составляют всего 16%. В эти белки вносили мутации, такие, что белки не теряли свою структуру. В результате, последовательно было получено несколько пар белков с высоким сходством последовательностей, но разными укладками (рис. 2, 3). У белков GA88 и GB88 одинаковые остатки составляют 88%, у GA95 и GB95 — 95%, у GA98 и GB98 — 98% (единственное их различие — аминокислотный остаток в 45 позиции: лейцин GA98 и тирозин GB98). Несмотря на вносимые мутации, белки сохраняют свою функцию: GA88, GA95, GA98 связывают сывороточный альбумин (GA98 также связывает IgG), GB88, GB95, GB98 связывают IgG [1-3]. A B Рисунок 1. (А) GA98 (Б) GВ98 α-спирали в GA98 и соответствующие а.о. в GВ98 раскрашены разными цветами. Показан единственный остаток, не совпадающий в GA98 и GВ98. GA GB GA88 GB88 TM=69.4˚C TM=57.5˚C GA95 GB95 TM=50.0˚C TM=48.7˚C GA98 GB98 TM=37.0˚C TM=35.0˚C Рисунок 2. Показаны изменения белков в эксперименте. Синим показаны аминокислотные остатки, идентичные аминокислотным остаткам в парном белке. Красным — различающиеся. Выравнивание показано на рисунке 3. Для мутированных белков приведена средняя точка тепловой денатурации (чем выше ее значение, тем белок стабильнее). (использованы данные [2]) Рисунок 3. Последовательности белков. Синим показаны аминокислотные остатки, идентичные аминокислотным остаткам в парном белке. Красным — различающиеся. На рисунке 4 показаны выравнивания белков, полученных на разных стадиях эксперимента. А Б Рисунок 4. (А) GA и его производные. (Б) GВ и его производные. Зеленым цветом показаны GA и GВ, голубым - GA88 и GВ88, красным - GA95 и GВ95, желтым - GA98 и GВ98. Каким образом белки, ушедшие так далеко от начальной аминокислотной последовательности, могут сохранять третичную структуру почти неизменной? Вероятно, дело в том, что остатки, составляющие гидрофобное ядро, в обеих структурах остаются практически неизменными (рис. 5) [1, 2]. A Б Рисунок 5. (А) Последовательности GA88 и GA95. Оранжевым отмечены остатки, составляющие гидрофобные ядра белков. Красным отмечены остатки, которые подвергаются мутации при переходе от GA88 к GA95. (Б) Последовательности GВ88 и GВ95. Фиолетовым отмечены остатки, составляющие гидрофобные ядра белков. Красным отмечены остатки, которые подвергаются мутации при переходе от GВ88 к GВ95. Вносимые мутации не затрагивают гидрофобные ядра белков. (использованы данные [1, 2]) GA98 и GВ98 различаются всего одним аминокислотным остатком. При внесении мутации L45Y в GA98, взаимодействия, стабилизировавшие 3α конформацию GA98, теряются, но появляются взаимодействия, стабилизирующие 4β+α конформацию GВ98. Взаимодействия 45ого лейцина, стабилизирующие 3α конформацию GA98, это гидрофобные взаимодействия с I33, I49, L32, Y29. Взаимодействия 45ого тирозина, стабилизирующие 4β+α конформацию GВ98: гидрофобное взаимодействие с ароматическим кольцом F52 и водородные связи с D47 и Y3 (рис. 6) [3]. Аминокислотные последовательности белков GA95 и GВ95 были предложены в качестве задания на CASP8. Большинство групп правильно предсказали структуру G В95, но только четыре группы смогли при этом правильно предсказать структуру G А95 (рис. 7) [4]. В эксперименте было показано изменение структуры белка в результате изменения аминокислотной последовательности. Возможно, подобные события происходят и в эволюции. На примере белков семейства Cro (транскрипционные факторы бактериофагов) можно наблюдать подобный процесс (рис. 8). Было показано, что пять белков семейства попарно имеют сходные последовательности. Была разрешена структура этих белков. Белки P22, Afe01, Xfaso 1 образованы тремя α-спиралямим, мономеры. Белки Pfl 6 и λ включают βлисты и α-спирали, димеры. При этом процент идентичных остатков в выравнивании Xfaso 1 и Pfl 6 - 45% [7]. A Б Рисунок 6. (А) Аминокислотные остатки, стабилизирующие 3α конформацию GA98. (Б) Аминокислотные остатки, стабилизирующие 4β+α конформацию GВ98. А Б Рисунок 7. (рис. из [5, 6]) (А) Global Distance Test для предсказаний структуры GA95. (Б) Global Distance Test для предсказаний структуры GВ95. Ось X: процент аминокислотных остатков в предсказанной структуре, Сα атомы которых отклоняются от структуры, полученной методом ЯМР не более чем на Сα DISTANCE cutoff. Ось Y: Сα DISTANCE cutoff (0.5A, 1.0A, 1.5A, ... , 10.0Å). Каждая линия — предсказание одной из групп. P22 E=0.008 ID=42% Afe01 E=1e-06 ID=39% Xfaso 1 E>10000 ID=25% E=0.049 ID=45% Pfl 6 E=5e-07 ID=43% λ Рисунок 8. Белки семейства Cro, принадлежащие различным фагам. Структура Afe01 была получена в низком разрешении и не представлена в банке PDB. Показаны значения E-value и процент идентичных остатков в выравниваниях пар белков. (использованы данные [7]) Список литературы 1. Yanan He, Yihong Chen, Patrick Alexander, Philip N. Bryan, and John Orban «NMR structures of two designed proteins with high sequence identity but different fold and function» PNAS 2008 105 (38) 14412-14417 2. Patrick A. Alexander, Yanan He, Yihong Chen, John Orban, and Philip N. Bryan «A minimal sequence code for switching protein structure and function» PNAS 2009 106 (50) 2114921154 3. Yanan He, Yihong Chen, Patrick A. Alexander, Philip N. Bryan, John Orban «Mutational Tipping Points for Switching Protein Folds and Functions» Structure 2012 20 (2) 283-291 4. Horst JA, Samudrala R. «Diversity of protein structures and difficulties in fold recognition: the curious case of protein G» F1000 Biology Reports. 2009 1:69 5. http://predictioncenter.org/casp8/gdtplot.cgi?target=T0498-D1&models=all&group= 6. http://predictioncenter.org/casp8/gdtplot.cgi?target=T0499-D1&models=all&group= 7. Christian G. Roessler, Branwen M. Hall, William J. Anderson, Wendy M. Ingram, Sue A. Roberts, William R. Montfort, and Matthew H. J. Cordes «Transitive homology-guided structural studies lead to discovery of Cro proteins with 40% sequence identity but different folds» PNAS 2008 105 (7) 2343-2348
